新(xīn)聞中(zhōng)心
來源: 江西立康(公(gōng)衆号)
2022年4月,T2T聯盟(國(guó)際科(kē)學(xué)團隊端粒到端粒聯盟)發表了首個完整的人類基因組。這一成就是通過實驗和計算上的一系列創新(xīn)實現的,而長(cháng)讀長(cháng)測序正是負責生成T2T數據的主要技(jì )術。因此,長(cháng)讀長(cháng)測序技(jì )術被《Nature Methods》雜志(zhì)評為(wèi)2022年最佳科(kē)學(xué)方法,正幫助全球科(kē)學(xué)家從人類及其他(tā)物(wù)種的基因組、轉錄組和表觀基因組中(zhōng)獲得大量發現。無論是PacBio還是Nanopore技(jì )術,在測序前,樣本的濃度和純度測量均可(kě)由NanoPhotometer®完成,結合完整性數據,以确保樣本質(zhì)量符合測序要求。

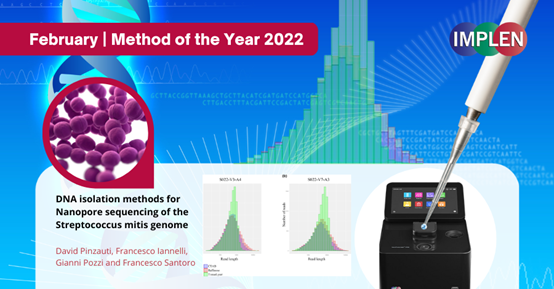
坐(zuò)标:意大利錫耶納大學(xué) 分(fēn)子微生物(wù)學(xué)與生物(wù)技(jì )術實驗室
期刊:《Microbial Genomics》
測序方法:Oxford Nanopore
牛津納米孔技(jì )術是細菌基因組學(xué)的重要工(gōng)具(jù),使實現長(cháng)讀長(cháng)測序可(kě)能(néng)。緩症鏈球菌是一種革蘭氏陽性細菌,屬于口腔共生微生物(wù)群。有(yǒu)時可(kě)能(néng)是感染性心内膜炎、菌血症和敗血症等疾病的病因。緩症鏈球菌基因組的重複性破壞了僅依靠短測序讀數的完整基因組的組裝(zhuāng)。牛津納米孔測序被證明通過産(chǎn)生長(cháng)讀長(cháng)信息來克服這一限制,從而能(néng)夠解析基因組重複區(qū)域并組裝(zhuāng)完整的基因組序列。由于納米孔測序運行的輸出受到基因組DNA質(zhì)量和分(fēn)子量的強烈影響,因此DNA分(fēn)離是優化測序運行的關鍵步驟。
研究小(xiǎo)組發表了三種DNA分(fēn)離方法的比較結果,這三種方法對高度重組的緩症鏈球菌基因組的測序進行了驗證,旨在從樣本中(zhōng)分(fēn)離出純高分(fēn)子量DNA,作(zuò)為(wèi)牛津納米孔測序實驗的模闆。研究結果表明,使用(yòng)基于機械裂解的方法分(fēn)離的DNA,盡管成本更低、速度更快,但不會産(chǎn)生超長(cháng)讀取(大讀取長(cháng)度為(wèi)59516個堿基),也不允許組裝(zhuāng)圓形完整基因組。使用(yòng)兩種基于酶解的方法分(fēn)離的DNA産(chǎn)生超長(cháng)讀取,高達181199個堿基,實現了循環完整基因組組裝(zhuāng)。這些方法可(kě)容易地應用(yòng)于從難裂解的革蘭氏陽性細菌中(zhōng)分(fēn)離高分(fēn)子量基因組DNA。
NanoPhotometer®應用(yòng):基因組DNA純度比值測量。
坐(zuò)标:美國(guó)明尼蘇達大學(xué) 動物(wù)科(kē)學(xué)系
期刊:《Nucleic Acids Research》
測序方法:Oxford Nanopore
接下來是克裏斯托弗·福爾克(Christopher Faulk)在NAR上發表的突破性文(wén)章,闡述了長(cháng)讀測序的時間和成本節約潛力。通常,參考動物(wù)基因組生成是一項耗時且成本高昂的操作(zuò)。使用(yòng)納米孔技(jì )術的DNA測序提供了直接、實時、長(cháng)讀取、可(kě)擴展、便攜式、自動化、快速和全面的基因組分(fēn)析。
Faulk僅在一周内就花(huā)費1000美元就對這一黑色木(mù)匠螞蟻的基因組進行了測序。與核基因組一起,線(xiàn)粒體(tǐ)基因組與線(xiàn)粒體(tǐ)基因組以及生活在螞蟻體(tǐ)内的兩種共生細菌一起組裝(zhuāng)。納米孔技(jì )術還使同一隻螞蟻的表觀遺傳學(xué)測量成為(wèi)可(kě)能(néng),并複制了其他(tā)顯示DNA甲基化程度很(hěn)低的研究。參考基因組在連續性和蛋白質(zhì)預測準确性方面優于其他(tā)螞蟻物(wù)種。這種方法将允許其他(tā)基礎型實驗室以低成本創建高質(zhì)量的基因組組件。
NanoPhotometer®應用(yòng):基因組DNA純度比值測量。

坐(zuò)标:印尼布拉幹薩理(lǐ)工(gōng)大學(xué) 農學(xué)和園藝系
期刊:《Data in Brief》
測序方法:PicBio
野肉豆蔻(Myristica fatua)是印度尼西亞的一種重要香料,研究人員正在對該物(wù)種的基因組資源進行分(fēn)析,發表了 Myristica fatua編碼序列(CDS),作(zuò)為(wèi)第一個轉錄組參考,該序列利用(yòng)牛津納米孔技(jì )術的長(cháng)讀測序獲得了全長(cháng)轉錄組組件。從這些數據中(zhōng)可(kě)以獲得全長(cháng)轉錄本,這有(yǒu)助于研究基因表達分(fēn)析。該信息為(wèi)與肉豆蔻屬相關屬的作(zuò)物(wù)育種計劃提供了EST微衛星分(fēn)子标記數據集,并可(kě)用(yòng)于鑒定與類黃酮生物(wù)合成相關的全長(cháng)轉錄物(wù),供分(fēn)子生物(wù)學(xué)家在肉豆蔻屬和相關屬的下遊分(fēn)析中(zhōng)使用(yòng)。
NanoPhotometer®應用(yòng):肉豆蔻葉中(zhōng)提取的RNA的定量和純度分(fēn)析。

坐(zuò)标:波多(duō)黎各大學(xué)馬亞圭斯分(fēn)校 生物(wù)學(xué)系
期刊:《Genes》
測序方法:PicBio
大安(ān)的列斯群島的亞馬遜鹦鹉,代表了島嶼上物(wù)種形成的一個模型,類似于加拉帕戈斯群島的達爾文(wén)雀。亞馬遜鹦鹉從中(zhōng)美洲大陸遷徙大安(ān)的列斯群島,但對于這種情況發生的方式和時間沒有(yǒu)達成共識。多(duō)國(guó)組成的研究小(xiǎo)組發表了一項研究,該研究對大安(ān)的列斯群島所有(yǒu)現存亞馬遜鹦鹉物(wù)種(a.leucocephala、a.agilis、a.claria、a.ventralis和a.vittata)進行了長(cháng)讀長(cháng)測序,并注釋了完整的線(xiàn)粒體(tǐ)基因組,包括注釋的線(xiàn)粒體(tǐ)基因組圖,種群多(duō)樣性和進化曆史。
這項研究的數據支持踏腳石擴散(stepping-stone dispersal)和物(wù)種形成假說,該假說描述了祖先種群從中(zhōng)美洲大陸抵達時,大約開始于3.47 MYA(百萬年前),并導緻了整個安(ān)的列斯群島物(wù)種的多(duō)樣化,到達波多(duō)黎各島在0.67 MYA。這一分(fēn)析有(yǒu)助于理(lǐ)解進化曆史,和對序列變化進行後續評估,并有(yǒu)助于設計未來的保護工(gōng)作(zuò)。
NanoPhotometer®應用(yòng):提取的基因組DNA濃度測量。

如果您使用(yòng)基因測序作(zuò)為(wèi)研究手段,可(kě)根據通量需求選擇多(duō)種型号的NanoPhotometer®作(zuò)為(wèi)對少量或大量樣本質(zhì)控的工(gōng)具(jù)。相信Implen能(néng)夠成為(wèi)您在基因測序工(gōng)作(zuò)中(zhōng)值得信賴的好幫手。哪裏有(yǒu)測序,哪裏就有(yǒu)Implen。
涉及的研究論文(wén)地址:
https://www.ncbi.nlm.nih.gov/pmc/articles/PMC8942023/
https://doi.org/10.1093/nar/gkac510
https://doi.org/10.1016/j.dib.2022.108838
https://doi.org/10.3390/genes12040608
